
计算生物学研究团队

【负责人介绍】
刘宇,博士,研究员,博士生导师。入选国家海外高层次人才引进计划青年项目,上海市东方学者岗位计划。本科毕业于南开大学生命科学学院,研究生就读于北京协和医学院,随后于美国儿童肿瘤专科排名第一的St. Jude Children’s Research Hospital计算生物学系完成博士后训练。现任1066vip威尼斯附属上海儿童医学中心儿科转化医学研究所PI。

【研究方向与成果】
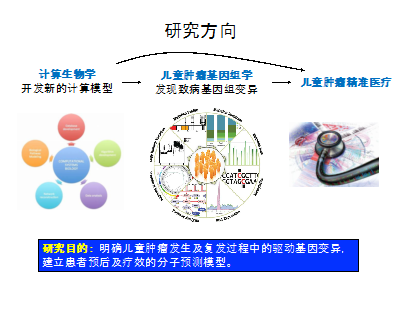
儿童肿瘤基因组学的计算与临床转化研究
课题组致力于儿童肿瘤基因组学研究,结合新一代的测序技术,开发计算生物学分析方法,整合分析多组学高通量测序数据研究儿童肿瘤基因组中的致病变异,并探索基因组学在临床诊疗中的转化应用。近年来作为主要完成人,(1)通过泛基因组分析(Pan-Cancer Analysis),绘制包括6种主要类型儿童肿瘤的体细胞变异图谱,发现多个新的驱动基因变异,从基因组水平明确儿童肿瘤不是成人肿瘤的缩微版,为儿童肿瘤的精准医疗奠定了理论基础和数据支撑(Nature 2018);(2)深入解析儿童T细胞急性淋巴细胞白血病基因组变异图谱,揭示了驱动基因变异间的协同作用是决定其生物学功能的关键(Nature Genetics 2017);(3)针对增强子、绝缘子等DNA调控区域基因组变异开发了新的计算生物学分析模型cis-X,实现了针对单一肿瘤基因组的非编码功能变异分析,突破了既往基因组研究局限于编码区的限制,进而发现新的肿瘤致病变异和基因,为肿瘤的精准诊疗提供新的靶点(Nature Genetics 2020,Cancer Discovery 2018);(4)基于基因组分析的临床转化研究,探索基因组分析应用于临床诊断及治疗方式的选择(Nature Communications 2016)。
【代表性论著】
Yu Liu#,* , C. Li#, S. Shen#, X. Chen, K. Szlachta, M. Edmonson, Y. Shao, X. Ma, J. Hyle, S. Wright, B. Ju, M. Rusch, Ya. Liu, B. Li, M.Macias, L. tian, J. Easton, M. Qian, J. Yang, S. Hu, A. T. Look, J. Zhang*. Discovery of regulatory noncoding variants in individual cancer genomes by using cis-X. Nature Genetics doi: 10.1038/s41588-020-0659-5. Online ahead of print (2020).
X. Ma#, Yu Liu#, Yanling Liu, L. B. Alexandrov, M. Edmonson, C. Gawad, X. Zhou, Y. Li, M. C. Rusch, J Easton, R. Huether, V. Gonzalez-Pena, M. Wilkinson, L. C. Hermida, S. Davis, E. Sioson, S. Pounds, X. Cao, R. E. Ries, Z. Wang, X. Chen, L. Dong, S. J. Diskin, M. A. Smith, J. M. G. Auvil, P. S. Meltzer, C. C. Lau, E. J. Perlman, J. Maris, S. Meshinchi, S. P. Hunger, D. S. Gerhard and J. Zhang*. Pan-cancer genome and transcriptome analyses of 1,699 pediatric leukemias and solid tumors. Nature 555, 371-376 (2018).
M. W. Zimmerman#, Y. Liu#, S. He, A. D. Durbin, B. J. Abraham, J. Easton, Y. Shao, B. Xu, S. Zhu, X. Zhang, Z. Li, N. Weichert-Leahey, R. A. Young*, J. Zhang* and A. T. Look*. MYC drives a subset of high-risk pediatric neuroblastomas and is activated through mechanisms including enhancer hijacking and focal enhancer amplification. Cancer Discovery 8, 320-335 (2018).
Y. Liu, J. Easton, Y. Shao, J. Maciaszek, Z Wang, M. R. Wilkinson, K. McCastlain, M. N. Edmonson, S. B. Pounds, L. Shi, X. Zhou, X. Ma, E. Sioson, Y. Li, M. Rusch, P. Gupta, D. Pei, C. Cheng, M. A. Smith, J. G. Auvil, D. S. Gerhard, M. V. Relling, N. J. Winick, A. J. Carroll, N. A. Heerema, E. Raetz, M. Devidas, C. L. Willman, R. C. Harvey, W. L. Carroll, K. P. Dunsmore, S. S. Winter, B. L. Wood, B. P. Sorrentino, J. R. Downing, M. L. Loh, S. P. Hunger*, J. Zhang* and C. G. Mullighan*. The genomic landscape of pediatric and young adult T-lineage acute lymphoblastic leukemia. Nature Genetics 49, 1211-1218 (2017).
Y. Liu#, J. Zhang#, L. Li#, G. Yin#, J. Zhang#, S. Zheng, H. Cheung, N. Wu, N. Lu, X. Mao, L. Yang, J. Zhang, L. Zhang, S. Seth, H. Chen, X. Song, K. Liu, Y. Xie, L. Zhou, C. Zhao, N. Han, W. Chen, S. Zhang, L. Chen, W. Cai, L. Li, M. Shen, N. Xu, S. Cheng, H. Yang, J. J. Lee, A. Correa, J. Fujimoto, C. Behrens, C. W. Chow, W. N. William, J. V. Heymach, W. K. Hong, S. Swisher, Wistuba, II, J. Wang, D. Lin, X. Liu, P. A. Futreal*, Y. Gao*. Genomic heterogeneity of multiple synchronous lung cancer. Nature Communications 7, 13200 (2016).
最新文章
更多- 血液肿瘤耐药复发研究团队 2021-10-13
- 出生缺陷遗传学研究团队 2021-10-13
- 儿童感染研究团队 2021-10-13
- 发育生物学研究团队 2021-10-13
- 儿童实体肿瘤基础和转化医学研究团队 2021-10-13
- 分子遗传研究团队 2021-10-13
- 骨髓微环境与血液疾病研究团队 2021-10-13
- 临床代谢与质谱代谢组学研究团队 2021-10-13
- 计算生物学研究团队 2021-10-13

